[Global Technology Breifings]
лӢЁл°ұм§Ҳ л””мһҗмқём—җ мқёкіөм§ҖлҠҘмқҙ мӮ¬мҡ©лҗҳлӢӨ
By Jin Sub Lee, NATURE COMPUTATIONAL SCIENCE, May 4, 2023
мқёкіөм§ҖлҠҘмқҖ мҶҢ비мһҗ мӨ‘мӢ¬ л°Ҹ мқјмғҒм Ғмқё 비мҰҲлӢҲмҠӨ м• н”ҢлҰ¬мјҖмқҙм…ҳмқ„ мһҗлҸҷнҷ”н•ҳлҠ” лҚ° мғҒлӢ№н•ң мҳҒн–Ҙмқ„ лҜём№ҳкІ м§Җл§Ң, ліөмһЎм„ұмңјлЎң мқён•ҙ мқёк°„мқҙ л°ңм „н• мҲҳ м—ҶлҠ” мҳҒм—ӯм—җм„ң к°ҖмһҘ нҒ° кё°м—¬лҘј н• кІғмқҙлӢӨ.
мқҙкІғмқҙ л°”лЎң лӢЁл°ұм§Ҳ л””мһҗмқём—җ мқёкіөм§ҖлҠҘмқ„ мӮ¬мҡ©н•ҳлҠ” кІғкіј кҙҖл Ёлҗң мғҲлЎңмҡҙ кІ°кіјк°Җ л§Өмҡ° нқҘлҜёлЎңмҡҙ мқҙмң мқҙлӢӨ.
вҖҳл„ӨмқҙмІҳ м»ҙн“Ён…Ңмқҙм…”л„җ мӮ¬мқҙм–ёмҠӨ(Nature Computational Science)вҖҷ м Җл„җмқҖ мөңк·ј нҶ лЎ нҶ лҢҖн•ҷм—җм„ң мқёкіөм§ҖлҠҘ мӢңмҠӨн…ңмқ„ мӮ¬мҡ©н•ҳм—¬ мһҗм—°м—җм„ң л°ңкІ¬лҗҳм§Җ м•ҠлҠ” лӢЁл°ұм§Ҳмқ„ мғқм„ұн•ҳлҠ” м—°кө¬ кІ°кіјлҘј л°ңн‘ңн–ҲлӢӨ.
мқҙ мқёкіөм§ҖлҠҘ мӢңмҠӨн…ңмқҖ к·ёлҰј мқёкіөм§ҖлҠҘ мҶҢн”„нҠёмӣЁм–ҙ лӢ¬лҰ¬(DALL-E)мҷҖ лҜёл“ңм ҖлӢҲ(Midjourney)мҷҖ к°ҷмқҖ мқёкё° мһҲлҠ” мқҙлҜём§Җ мғқм„ұ н”Ңлһ«нҸјкіј лҸҷмқјн•ң кё°мҲ мқё мғқм„ұ нҷ•мӮ°мқ„ мӮ¬мҡ©н•ңлӢӨ.
мқҙ мӢңмҠӨн…ңмқҖ мҷ„м „нһҲ мғҲлЎңмҡҙ м№ҳлЈҢмҡ© лӢЁл°ұм§Ҳмқ„ ліҙлӢӨ нҡЁмңЁм Ғмқҙкі мң м—°н•ҳкІҢ к°ңл°ңн•ҳлҠ” мҶҚлҸ„лҘј лҶ’мқј кІғмқ„ мҳҲкі н•ҳкі мһҲлӢӨ.
мҰү, мқҙ лӘЁлҚёмқҖ мқҙлҜём§Җ н‘ңнҳ„мқ„ мӢңмһ‘мңјлЎң л§Өмҡ° л№ лҘё мҶҚлҸ„лЎң вҖҳмҷ„м „нһҲ мғҲлЎңмҡҙвҖҷ лӢЁл°ұм§Ҳмқ„ мғқм„ұн•ҳлҠ” л°©лІ•мқ„ н•ҷмҠөн•ңлӢӨ.
к·ёлҰ¬кі мқҙл ҮкІҢ мғқм„ұн•ҳлҠ” лӘЁл“ лӢЁл°ұм§ҲмқҖ мғқл¬ј л¬јлҰ¬н•ҷм ҒмңјлЎң мӢӨм ңм Ғмқё кІғмІҳлҹј ліҙмқёлӢӨ. мҰү, м„ёнҸ¬ лӮҙм—җм„ң нҠ№м • кё°лҠҘмқ„ мҲҳн–үн• мҲҳ мһҲлҠ” кө¬м„ұмңјлЎң м ‘нҳҖ мһҲлӢӨ(folding)лҠ” мқҳлҜёмқҙлӢӨ.
лӢЁл°ұм§ҲмқҖ 3м°Ёмӣҗ лӘЁм–‘мңјлЎң м ‘нһҲлҠ” м•„лҜёл…ёмӮ° мӮ¬мҠ¬лЎң л§Ңл“Өм–ҙм§Җл©°, мқҙлҠ” лӢӨмӢң лӢЁл°ұм§Ҳ кё°лҠҘмқ„ кІ°м •н•ңлӢӨ.
кё°мЎҙ лӢЁл°ұм§Ҳмқҙ м–ҙл–»кІҢ м ‘нһҲлҠ”м§Җ лҚ” мһҳ мқҙн•ҙн•ҳл©ҙм„ң м—°кө¬мһҗл“ӨмқҖ мһҗм—°м—җм„ңлҠ” мғқм„ұлҗҳм§Җ м•ҠлҠ” м ‘нһҳ нҢЁн„ҙмқ„ м„Өкі„н•ҳкё° мӢңмһ‘н–ҲлӢӨ.
к·ёлҹ¬лӮҳ к°ҖмһҘ нҒ° лҸ„м „мқҖ к°ҖлҠҘн•ҳкі кё°лҠҘм Ғмқё м ‘нһҳмқ„ мғҒмғҒн•ҳлҠ” кІғмқҙм—ҲлӢӨ. м–ҙл–Ө м ‘нһҳмқҙ мӢӨм ң лӢЁл°ұм§Ҳ кө¬мЎ°м—җм„ң мһ‘лҸҷн•ҳлҠ”м§Җ мҳҲмёЎн•ҳлҠ” кІғмқҖ л§Өмҡ° м–ҙл өлӢӨ.
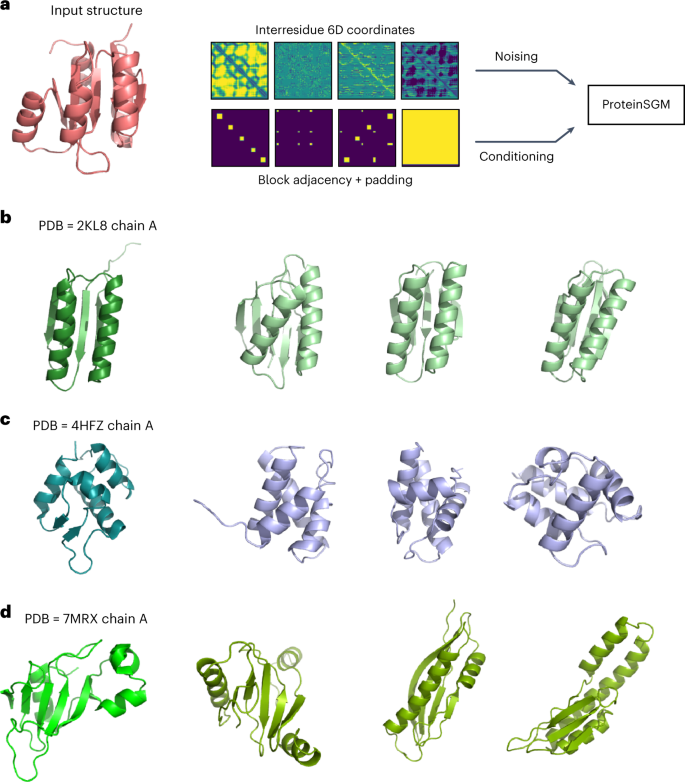
н•ҳм§Җл§Ң м—°кө¬мһҗл“ӨмқҖ лӢЁл°ұм§Ҳ кө¬мЎ°мқҳ мғқл¬ј л¬јлҰ¬н•ҷ кё°л°ҳ н‘ңнҳ„кіј мқҙлҜём§Җ мғқм„ұ кіөк°„мқҳ нҷ•мӮ° л°©лІ•мқ„ кІ°н•©н•ЁмңјлЎңмҚЁ мқҙ л¬ём ңлҘј н•ҙкІ°н•ҳкё° мӢңмһ‘н–ҲлӢӨ.
м—°кө¬мһҗл“Өмқҙ н”„лЎңн…ҢмқёSGM(ProteinSGM)мңјлЎң л¶ҖлҘҙлҠ” мғҲлЎңмҡҙ мӢңмҠӨн…ңмқҖ кё°мЎҙ лӢЁл°ұм§Ҳмқҳ мқҙлҜём§ҖмҷҖ мң мӮ¬н•ң н‘ңнҳ„мқ„ лҢҖлҹүмңјлЎң лҒҢм–ҙмҷҖм„ң, к·ё кө¬мЎ°лҘј м •нҷ•н•ҳкІҢ мқёмҪ”л”©н•ңлӢӨ.
м—°кө¬мһҗл“ӨмқҖ мқҙлҹ¬н•ң мқҙлҜём§ҖлҘј мғқм„ұ нҷ•мӮ° лӘЁлҚём—җ мһ…л Ҙн•ҳм—¬ к°Ғ мқҙлҜём§Җк°Җ лӘЁл‘җ л…ёмқҙмҰҲк°Җ лҗ л•Ңк№Ңм§Җ м җм°Ём ҒмңјлЎң л…ёмқҙмҰҲлҘј 추к°Җн•ңлӢӨ.
мқҙ лӘЁлҚёмқҖ мқҙлҜём§Җм—җ л…ёмқҙмҰҲк°Җ м–ҙл–»кІҢ мҰқк°Җн•ҳлҠ”м§Җ 추м Ғн•ң нӣ„, н”„лЎңм„ёмҠӨлҘј м—ӯмңјлЎң мӢӨн–үн•ҳм—¬ л¬ҙмһ‘мң„ н”Ҫм…Җмқ„ мҷ„м „нһҲ мғҲлЎңмҡҙ лӢЁл°ұм§Ҳм—җ н•ҙлӢ№н•ҳлҠ” м„ лӘ…н•ң мқҙлҜём§ҖлЎң ліҖнҷҳн•ҳлҠ” л°©лІ•мқ„ н•ҷмҠөн•ңлӢӨ.
мғҲлЎңмҡҙ лӢЁл°ұм§Ҳмқ„ н…ҢмҠӨнҠён•ҳкё° мң„н•ҙ м—°кө¬мһҗл“ӨмқҖ лЁјм Җ л”Ҙл§Ҳмқёл“ң(DeepMind) мҶҢн”„нҠёмӣЁм–ҙ м•ҢнҢҢнҸҙл“ң 2мқҳ к°ңм„ лҗң лІ„м „мқё мҳӨл©”к°ҖнҸҙл“ң(OmegaFold)лҘј м„ нғқн–ҲлӢӨ.
л‘җ н”Ңлһ«нҸј лӘЁл‘җ мқёкіөм§ҖлҠҘмқ„ нҷңмҡ©н•ҳм—¬ м•„лҜёл…ёмӮ° м„ңм—ҙмқ„ кё°л°ҳмңјлЎң лӢЁл°ұм§Ҳ кө¬мЎ°лҘј мҳҲмёЎн•ңлӢӨ.
мҳӨл©”к°ҖнҸҙл“ңлҘј нҶөн•ҙ м—°кө¬мһҗл“ӨмқҖ кұ°мқҳ лӘЁл“ мғҲлЎңмҡҙ м„ңм—ҙл“Өмқҙ к·ёл“Өмқҙ мӣҗн•ҳлҠ” лӢЁл°ұм§Ҳ кө¬мЎ°лЎң м ‘нһҲлҠ” кІғмқ„ нҷ•мқён–ҲлӢӨ.
мқҙнӣ„ м—°кө¬мһҗл“ӨмқҖ мӢңн—ҳкҙҖ мӢңн—ҳмқ„ нҶөн•ҙ, к·ё кө¬мЎ°к°Җ лӢЁмҲңн•ң нҷ”н•ҷ нҷ”н•©л¬јмқҳ лҒҲмқҙ м•„лӢҢ лӢЁл°ұм§Ҳмһ„мқ„ нҷ•мқён–ҲлӢӨ.
мҳӨл©”к°ҖнҸҙл“ңмҷҖмқҳ л§Өм№ӯкіј мӢӨн—ҳмӢӨм—җм„ңмқҳ мӢӨн—ҳ н…ҢмҠӨнҠёлҘј нҶөн•ҙ м—°кө¬мһҗл“ӨмқҖ мқҙл“Өмқҙ м Ғм Ҳн•ҳкІҢ м ‘нһҢ лӢЁл°ұм§Ҳмһ„мқ„ нҷ•мӢ н• мҲҳ мһҲм—ҲлӢӨ.
мқҙл“ӨмқҖ мһҗм—° м–ҙл””м—җлҸ„ мЎҙмһ¬н•ҳм§Җ м•ҠлҠ” мҷ„м „нһҲ мғҲлЎңмҡҙ лӢЁл°ұм§Ҳ м ‘нһҳмқҙ нҷ•мқёлҗҳлҠ” кІғмқ„ ліҙкі лҶҖлһ„ мҲҳл°–м—җ м—Ҷм—ҲлӢӨ.
мқҙ м—°кө¬лҘј кё°л°ҳмңјлЎң, лӢӨмқҢ лӢЁкі„лҠ” м№ҳлЈҢ к°ҖлҠҘм„ұмқҙ к°ҖмһҘ лҶ’мқҖ н•ӯмІҙ л°Ҹ кё°нғҖ лӢЁл°ұм§Ҳм—җ лҢҖн•ң к°ңл°ңмқҙлӢӨ. мқҙ м—°кө¬мҷҖ к·ё кІ°кіјлҠ” м—°кө¬мһҗл“Ө лҝҗл§Ң м•„лӢҲлқј кҙҖкі„мһҗ, кҙҖкі„кё°м—… л“ұм—җлҸ„ л§Өмҡ° нқҘлҜёлЎңмҡҙ кІғмқҙ лҗ кІғмқҙлӢӨ.
- NATURE COMPUTATIONAL SCIENCE, May 4, 2023, вҖңScore-Based Generative Modeling for De Novo Protein Design,вҖқ by Jin Sub Lee, et al. © 2023 Springer Nature Limited. All rights reserved.
To view or purchase this article, please visit:
[Global Technology Breifings]
Score-Based Generative Modeling for De Novo Protein Design
By Jin Sub Lee, NATURE COMPUTATIONAL SCIENCE, May 4, 2023
While artificial intelligence will make a substantial impact in automating consumer oriented and everyday business applications, its greatest contribution will be in areas where humans are unable to make progress because of complexity.
ThatвҖҷs why new results related to using AI in protein design are so exciting.
The journal Nature Computational Science just published the results of research at the University of Toronto into using an artificial intelligence system to create proteins not found in nature.
This AI system uses generative diffusion, the same technology behind popular image-creation platforms such as DALL-E and Midjourney.
The system promises to speed drug developtirely new therapeutic proteins more efficient and flexible. The model learns to generate вҖңfully newвҖқ proteins, at a very high rate, starting from image representations.
And all the proteins it generates appear to be biophysically real, meaning they fold into configurations that enable them to carry out specific functions within cells.
Proteins are made from chains of amino acids that fold into three-dimensional shapes, which in turn dictate protein function.
With a better understanding of how existing proteins fold, researchers have begun to design folding patterns not produced in nature.
But a major challenge has been to imagine folds that are both possible and functional. ItвҖҷs been very hard to predict which folds will be real and work in a protein structure.
By combining biophysics-based representations of protein structure with diffusion methods from the image generation space, the researchers have begun to address this problem.
The new system, which the researchers call ProteinSGM, draws from a large set of image-like representations of existing proteins that encode their structure accurately.
The researchers feed these images into a generative diffusion model, which gradually adds noise until each image becomes all noise.
The model tracks how the images become noisier and then runs the process in reverse, learning how to transform random pixels into clear images that correspond to fully novel proteins.
To test their new proteins, the researchers first turned to OmegaFold, an improved version of DeepMindвҖҷs software AlphaFold 2.
Both platforms use AI to predict the structure of proteins based on amino acid sequences.
With OmegaFold, the team confirmed that almost all their novel sequences fold into the desired protein structures.
They then chose a smaller number to create physically in test tubes, to confirm the structures were proteins and not just stray strings of chemical compounds.
With matches in OmegaFold and experimental testing in the lab, they could be confident these were properly folded proteins.
They were amazed to see validation of these fully new protein folds that donвҖҷt exist anywhere in nature.
Next steps based on this work include further development of ProteinSGM for antibodies and other proteins with the most therapeutic potential.
This will be a very exciting area for research and entrepreneurship.
- NATURE COMPUTATIONAL SCIENCE, May 4, 2023, вҖңScore-Based Generative Modeling for De Novo Protein Design,вҖқ by Jin Sub Lee, et al. © 2023 Springer Nature Limited. All rights reserved.
To view or purchase this article, please visit: